
『【RStudio】RStudioを使う』シリーズでは、RStudioを使ってデータの統計処理、グラフの描画をしていきます。
第十二弾の今回は、#1〜#11で書いてきたコードの総まとめをしましょう。
目指すグラフの形はこちらに記載しています。
「RStudioを使う」シリーズは、生命科学系の統計処理に必要な基本的なR言語の扱い方をまとめていきます。
RStudioのインストール方法はこちらをご覧ください。
この記事では、細胞の核を染めた写真をImageJで解析した結果を比較していくのを目的としています。
その第一歩として、手動で計数した結果とImageJマクロを使って計数した結果に差があるのかどうか?をRを使って検定します。
ImageJマクロで出力した結果をdplyrを使ってまとめる
ImageJのマクロで核を数えるコードを書きましたが、一つの画像に対して一つのcsvファイルが出力されます。
そこで、RStudioの"dplyr"パッケージを使って計数結果をまとめて行きました。
ggplot2でグラフを描く
まとめたコードを元に"ggplot2"パッケージを使ってグラフを描画してきました。
ggsignifで検定結果を記入する
最後に"ggsignif"パッケージを使って検定結果を記入します。
Rで対応のあるt検定をするのはこちら
グラフに検定結果を記入する方法はこちら
コードの全体像を見る
では、今回のシリーズで書いたコードの全体像を見てみましょう。
|
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46 47 48 49 50 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 |
###今回使うパッケージを呼び出す### library(stringr) library(dplyr) library(ggplot2) library(ggsignif) ###ImageJ macroで解析した結果が入っているディレクトリを選ぶ### filelist <- list.files(pattern = ".csv") condition <-c() sample <- c() count <- c() for (i in 1:length(filelist)){ data <- read.csv(filelist[i]) filename <- str_split(filelist[i], "_", simplify = T) samplen <- length(filename)-2 condition <- c(condition, filename[1, 1]) sample <- c(sample, filename[1,samplen]) count <- c(count, (data[1,2])) } ###macroの結果をまとめたデータフレームを作る### macro <- data.frame(condition = condition, sample = sample, count = count) macro <- macro %>% mutate(method = rep("macro", nrow(macro))) ###手動で計数した結果が入っているディレクトリを選ぶ### manu <- read.csv("count.csv") manu <- manu %>% mutate(method = rep("manu", nrow(manu))) ###対応のあるt検定をする### t.test(macro$count, manu$count, paired = T) #コントロールのデータだけを集める cmacro <- macro %>% filter(condition == "control") cmanu <- manu %>% filter(condition == "control") #薬剤投与群のデータだけを集める tmacro <- macro %>% filter(condition == "treated") tmanu <- manu %>% filter(condition == "treated") #t検定の結果のp値を取り出す pval <- t.test(macro$count, manu$count, paired = T)[["p.value"]] cpval <- t.test(cmacro$count, cmanu$count, paired = T)[["p.value"]] cpval <- paste0("p = ", round(cpval, digits = 3)) #値を丸める tpval <- t.test(tmacro$count, tmanu$count, paired = T)[["p.value"]] tpval <- paste0("p = ", round(tpval, digits = 3)) #値をまとめる ###ggplot2を使ってグラフを描く### #グラフを書くためにmacroと手動の結果を一つにまとめる df <- bind_rows(macro, manu) p1 <- ggplot(data = df, aes(x = method, y = count, color = sample, group = sample)) p1 <- p1 + geom_line()+ geom_point(size = 3)+ facet_wrap(~condition)+ scale_color_viridis_d(option = "viridis") p1 #ggsignifの結果を反映する p2 <- p1 + geom_signif(comparisons = list(c("macro", "manu")), test = "t.test", #map_signif_level = TRUE, test.args = list(paired = T), y_position = max(df$count)*1.1, color = "black") p2 |
マクロのデータとマニュアルのデータが別々のフォルダに入っているので、ディレクトリを選ぶときに注意が必要です。
"p1"を実行すると
"p2"を実行すると
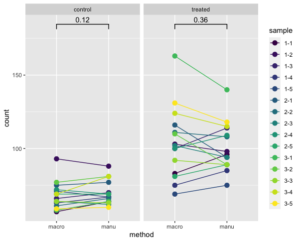
が表示されます。




























