
大学の講義で習ったはずなのに、突然、研究室での会話で出てきて「なんだっけ?」ってなる言葉ってありますよね。
講義で習ったことを全部覚えられるわけでもないので、日常的に使う言葉しかわからなくなってしまいます。
何回も繰り返して「なんだっけ?」を繰り返すことで、言葉の意味を少しずつ理解できるようになってくるのです。
さて、今回は「ORF(Open Reading Frame)」。
大学の講義(生命科学系なら)で絶対やったことはあるし、日常的に耳にする言葉でもあるのではないでしょうか?
いざ、説明してとなったら「なんだっけ?」となる言葉でもあるのではないかと思います。
「ORF」ってなに?
「ORF」とは、Open Reading Frameの略。
直訳すると、「開いた読み枠」という意味ですね。
一体何が「開い」ているんでしょう。
まずはDNAからタンパク質が合成される過程、「セントラルドグマ」について軽く復習して行きます。
「セントラルドグマ」とは
「セントラルドグマ(Central dogma)」とは、分子生物学の基本原則です。
生物の遺伝情報は、ゲノムDNA→(複製)→DNA→(転写)→RNA→(翻訳)→タンパク質の順に一方向に情報が流れて行くというもの。
(ウィルスなど一部例外がありますが基本的にはこの原則に則って情報が流れて行きます)

6種類の読み枠がある
mRNAからタンパク質が翻訳される時に、3つの塩基で一つのアミノ酸が指定されます。
この3つの塩基のことを「コドン」と呼びます。
(高校の教科書に「コドン表」が載っていますよね)
この「コドン」の並び方(読み枠)は、一つの塩基配列で6種類想定されてしまうのです。
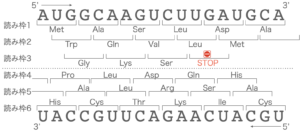
どの読み枠を使ってタンパク質を合成して行くのか、6種類もあったらわからなくなってしまいますよね。
そこで、タンパク質を合成する始まりの合図(開始コドン)が「AUG(メチオニン)」となっています。
終わりの合図も存在します。終止コドンと言って、「UAA、UGA、UAG」の3つあります。
(ちなみに私は「うあー(UAA)、うぐあー(UGA)、うあぐー(UAG)」と終止コドンを覚えました)
この開始コドンと終止コドンに挟まれた領域のことを「ORF」と呼びます。

あえて、曖昧に書きました。
もう少しだけ掘り下げてみましょう。
「遺伝子」と「ORF」の違い
ここまで見てきて、「遺伝子」と「ORF」ってほとんど一緒じゃないか?って思いませんでしたか?
この二つの使い分けをどうしたら良いのか、少し考えてみましょう。
そもそも「遺伝子」とは?
分子生物学の中でも「遺伝子」の定義にかなりの幅があります。
一番狭い解釈としては、
「タンパク質として翻訳されるmRNAの情報を含む核酸(DNA)上の特定の領域」
となり、構造遺伝子(シストロン)とも言われます。
転写因子が結合したり、転写量を制御(プロモーター、エンハンサー)したりするのも、DNA上の情報を読み取るには不可欠ですよね。
なので、もう少し広い解釈としては「シストロンに隣接する転写調節領域を含むDNAの領域」としてオペロンと呼ばれます。
しかし、DNAに書かれている情報は全てがmRNAに転写されて翻訳されるわけではなく、タンパク質を作るために必要なtransfer RNA(tRNA)やribosmomal RNA(rRNA)、RNA自体で転写し制御を行うノンコーディングRNAなどの情報も含まれています。
このような機能的なRNAのコード領域も「遺伝子」に含まれることもあります。
と、見てみると「遺伝子」の定義にも狭義と広義があって大分幅があるものみたいですね。
「ORF」との違いは?
実は膨大なDNA情報の中で「遺伝子」として機能がわかっているものはほんのわずかで、ほとんどが機能がわかっていません。
機能がわかっていないDNA配列のどこに「遺伝子(主にシストロン)」があるのかを探索する「遺伝子予測(gene prediction)」をするときに「遺伝子」ではなく、「ORF」という考え方が役に立ちます。
真核生物の遺伝子予測をする場合は転写されたmRNAを中心に考えます。
転写されて、スプライシングを受けたmRNAの「開始コドン(AUG)」から「終止コドン」までの領域はタンパク質に合成される可能性が高い配列です。
mRNA上の「開始コドン(AUG)」から「終止コドン」までの領域を探索して行くことで「遺伝子(シストトロン)」を見つけて行きます。
統一された定義があったほうが探索しやすいということで、「開始コドン(AUG)」から「終止コドン」までの領域を「ORF」と呼んでいます。
※ 原核生物のようにエクソンとイントロンが分かれていない場合は、DNA配列とORFは同じになります
イメージ的にはこんな感じでしょうか?
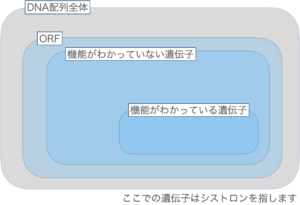
生物は常に例外があるもので、必ずしも「AUG」から翻訳が始まらなかったり、タンパク質ではなく小さなRNAが機能を持っていたりします。
そんな場合は「ORF」ではなく、別の基準が必要なのかもしれません。
生物ってまだわからないことが沢山あるんだなと感じますね。
まとめ
- 遺伝子予測の分野ではmRNA上の「開始コドン」から「終止コドン」までの領域をORFと言う
- ORFであっても機能的な役割(タンパク質が作られる、機能を持つRNAをコードしている)とは限らない
- 機能的があるとしてもORFの定義に当てはまるとは限らない
- 原核生物の場合、mRNA上のORF配列とDNAの配列は一緒になる
参考文献
酵母とシステムバイオロジーシリーズ 過剰発現・第8回「遺伝子・ORFはどのように決められるか?」














