
『【RStudio】RStudioを使う』シリーズでは、RStudioを使ってデータの統計処理、グラフの描画をしていきます。
第八弾の今回は、「t検定」をしていきます。
第七弾で加工したデータフレームを使って「t検定」をして行きましょう。
第二弾で書いたフローチャートの「ココ!」の部分です。

RStudioのインストール方法はこちらをご覧ください。
この記事では、細胞の核を染めた写真をImageJで解析した結果を比較していくのを目的としています。
その第一歩として、手動で計数した結果とImageJマクロを使って計数した結果に差があるのかどうか?をRを使って検定します。
検定をするためのデータを準備する
手動で計数したcsvデータを読み込む
これからマクロのデータと手動のデータを比較検定するので、手動で解析したcsvデータを読み込みましょう。
RStudioを使ってcsvデータ読み込む方法はこちらの記事にまとめています。
csvファイルを読み込む時には"read.csv"関数を使います。
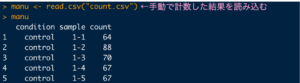
手動で解析した結果を"manu"という変数に入れました。
データを加工する
次に、t検定をしていくために必要なデータを集めるようにデータを加工していきます。
control群同士、treated群同士を比較するために、conditionごとにデータを抽出して見ましょう。
"tidyverse"パッケージの"dplyr"の"filter"関数を使って行きます。
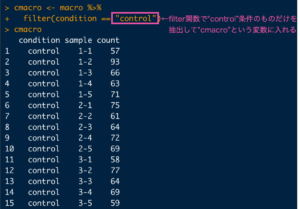
マクロで解析したデータのうち、control群のデータを抽出したものを"cmacro"という変数に入れました。
同様に、マクロで解析したtreated群のデータを抽出したもの、手動で解析したうちのcontrol群(cmanu)、treated群(tmanu)を抽出したものを作ります。
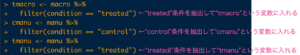
データの準備ができたので、t検定をして行きます。
今回は下の表の①〜③のP値を算出して行こうと思います。(セルの中は変数の名前です)
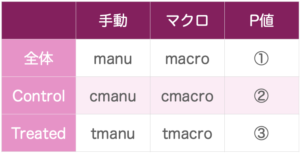
全体で比較したものを①、control群を比較したものを②、treated群を比較したものを③として、算出して行きましょう。
RStudioで対応のあるt検定をする
先程の表に従ってt検定をして行きましょう。
今回は、手動で数えたものとマクロを使って数えたものを比較して行きます。同じ写真を数えた結果を比較するので行う検定は、対応のあるt検定となります。
t検定についてはこちらにもまとめています。
RでT検定をする関数は、"t.test"です。

実際に"t.test"関数を使って見ましょう。
まずは表の①の全体の比較をして行きます。
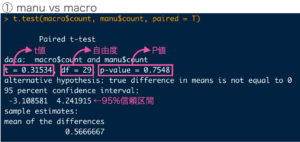
表に結果を反映すると、このような感じです。
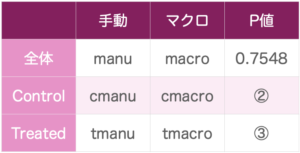
同じように②、③のデータも比較して行きます。
②はcontrol群同士を比較して行きます。使うデータは先程"filter"関数を使って加工したデータを使います。
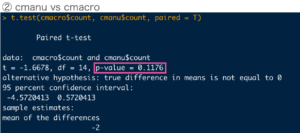
③はtreated群同士を比較して行きます。
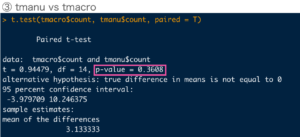
ピンクで囲った部分がp値です。結果を表に反映して行きましょう。
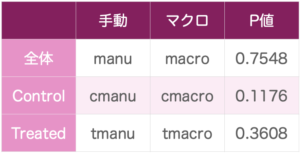
表が埋まりました。
t検定の結果(p値)を取り出す
検定ができましたが、これからグラフを描いていくにあたってp値をR上で取り出しておきたいと思います。
検定結果を取り出す方法を簡単にまとめます。
まずt検定を変数に入れてみて、その中身を見て見ましょう。
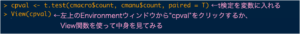
"View"関数を使って変数の中身を見ることができます。
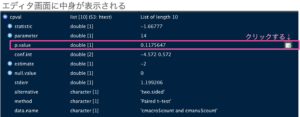
エディタ画面に表示された中身の中で、p値が書いてあるところをクリックしてみます。
![]()
添字をつければ中身を取り出せることがわかりました。
添字をつける手順で、①〜③のp値をそれぞれの変数に入れておきます。
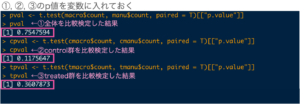
これは、この後グラフを書く時に使って行きます。



















