
『【RStudio】RStudioを使う』シリーズでは、RStudioを使ってデータの統計処理、グラフの描画をしていきます。
第11弾の今回は、"ggplot2"で描いたグラフに検定結果を反映させて行きます。
前回書いたグラフに足して行きましょう。
第二弾で書いたフローチャートの「ココ!」の部分です。

RStudioのインストール方法はこちらをご覧ください。
この記事では、細胞の核を染めた写真をImageJで解析した結果を比較していくのを目的としています。
その第一歩として、手動で計数した結果とImageJマクロを使って計数した結果に差があるのかどうか?をRを使って検定します。
"ggsignif"をインストールする
前回までに、ggplot2でグラフを描いてきました。
ggplot2に検定結果を反映させる方法は、自分で書き込む方法と、"ggsignif"を使って書き込む方法があります。
自分で書き込むとなるとかなりめんどくさいので、今回は"ggsignif"を使って書き込んでいきましょう。
"ggsignif"とは
"ggsignif"とはggplot2と互換性のあるパッケージで、ggplot2で描いたグラフの上に検定結果を反映するレイヤーを一枚乗せることができます。
詳しい使い方は、こちらのサイトをご覧ください。
使い始める前に"ggsignif"パッケージをインストールしましょう。
"ggsignif"はCRANで公式に配布されているパッケージなので、"install.packages()"でインストールできます。
"ggsignif"を使って検定結果をグラフに反映する
"ggsignif"を使ってggplot2で描いたグラフの上に検定結果を反映させて行きます。
ggplot2はレイヤーを一枚一枚重ねてグラフを描いていくイメージです。
"+"記号を使って、検定結果を反映するコードを書いて行きましょう。
その時に忘れてはいけないのは、"library(ggsignif)"でパッケージを呼び出しておくことです。
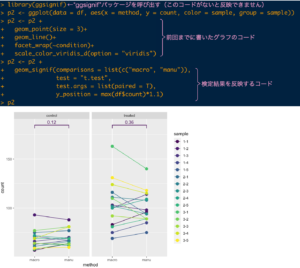
グラフの上に、検定結果が反映されました。
"geom_signif"の中身を詳しく見てみましょう。
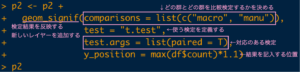
"geom_signif"を使うときに必要な引数としては、
- "comparisons":どの群とどの群を比較するかをリスト形式で指定します
- "test":どの検定を使うかを指定します(デフォルトではWelchの対応のないt検定が設定されています
- "test.args":対応のある検定の場合は"paired = TRUE"としておきます
追加で、"y_position"でどの高さに検定結果を表示させるかを指定しています。
"geom_signif"で出力された値と、事前に検定した結果が合っているのかを確認してみましょう。
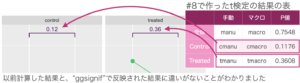
数値は丸くなっていますが、結果に違いがないことが確かめられました。
「Rで対応のあるt検定をする」の記事はこちら↓
検定結果をスターの形で表示させる
検定結果のp値が数値の形で表示されています。
コードを一行加えるだけで、スターをつけることができるので、最後にご紹介します。
"geom_signif"の引数に"map_signif_level = TRUE"を加えるだけ。
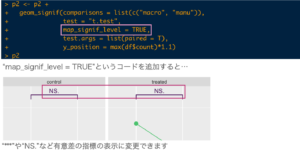
p < 0.05の時は"*"、p < 0.01の時は"**"、p < 0.001の時は"***"が付くようになっています。
検定結果を反映できるようになりました。
次回は、グラフを描くために必要なコードの総まとめをしようと思います。
自分で計算した検定結果を記入したい場合は、こちらの記事をご覧ください。



















