
「細胞を数えたいとき」ってどうしていますか?
核をDAPIやHoechstなどで染色して蛍光顕微鏡で撮影して、解析するのが一般的なのではないでしょうか?
生命科学系で画像解析といえば、ImageJですよね。
ImageJを使って細胞を計数する方法を、何回かに分けてまとめていきます。
ImageJをまだインストールしていない方は、こちらの記事でインストール方法をご紹介しています。
解析する戦略が定まったので、数回に分けてマクロを組んでいきます。
今回は、第一回として解析手順をマクロに記録するまでをまとめました。
ImageJを使って核を計数する流れ
今までの戦略をもう一度、振り返っていきます。
DAPIを使って染色した画像を使って、核を数えていました。
まずは、手動で数えてみてどれくらい時間がかかるのかを測ってみました。
次に、ImageJを使って解析する方法を模索していきます。
ImageJが「核」を認識するためには、「輝度閾値」を設定するのが良いことがわかりました。
「輝度閾値」で領域を指定しても、核が重なり合っている場合に一つになってしまいました。
ImageJに元々搭載されているWatershedを使ってみたのですが、うまく分割できず...
Watershedのパラメータを調整できるプラグインを導入してみました。
Adjustable Watershedを使ってみましたが、核を数えるのにはあまり向いてなさそう...
Interactive Watershedは割と使いやすいのでは無いかという手応え。
Interactive Watershedを使って、核が重なっている部分を分割することにしました。
解析の流れは、
- 画像を読み込む
- 8-bit画像に変換する
- Interactive Watershedでパラメータを設定して分割
- 分割された画像をAnalyze Particles...で計数
- 計数結果をcsvファイルに書き出す
となります。
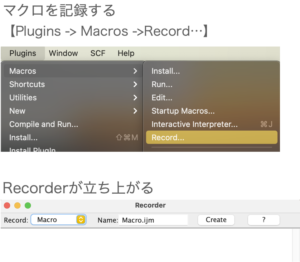
ImageJでマクロを記録する
解析の手順が決まってきたので、ImageJでその手順を追ってマクロとして記録していきましょう。
マクロは下記の手順で記録できます。
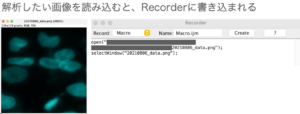
解析手順を踏んでいきます。
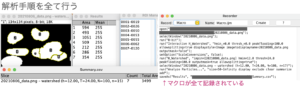
解析手順を全部踏むと、Recorderに全てのマクロが記録されています。
記録したマクロを使えるようにしていく
Recorderの“Create”をクリックすると記録したマクロを編集できるようになります。
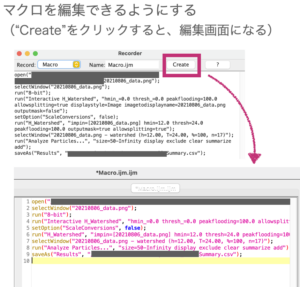
黄色い文字が関数名、ピンクの文字が変数を表しています。
関数と変数についてはこちらの記事をご覧ください。

とりあえず記録してできたマクロを何も考えずに、動かしてみましょう。
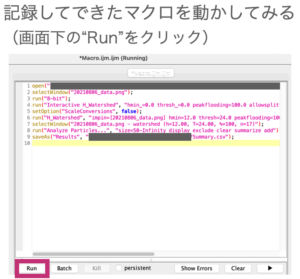
しかし...
マクロのエラーが出てしまいました...
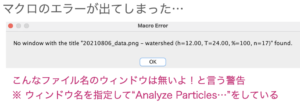
7行目の"selectWindow"から8行目の"run("Analyze Particles...")"の操作で、ウィンドウ名を指定して解析作業をしているのですが、そんな名前のウィンドウは無いよと言うエラーが出ました。
次回からは、記録して作ったマクロを編集して使えるようにしていきます。





















