
「細胞を数えたいとき」ってどうしていますか?
核をDAPIやHoechstなどで染色して蛍光顕微鏡で撮影して、解析するのが一般的なのではないでしょうか?
生命科学系で画像解析といえば、ImageJですよね。
ImageJを使って細胞を計数する方法を、何回かに分けてまとめていきます。
この記事では、人の目とコンピュータの画像の認識の違いについてまとめていきます。
ImageJの核の見つけ方と人の核の見つけ方
自分の目で見る場合、撮影した画像で「細胞はどこか?」「核はどこにあるか?」はほとんど何も考えずに区別できますよね。
「核を数える」という作業も、淡々とこなすことができます。
「人の目」は無意識に対象物(ここでは細胞の核)を特定することができる優れものなのです。
でも解析する画像が数10枚、数100枚と増えていくと、さすがに手で数えるのは時間の無駄。
30枚の画像を手で数えた場合の時間はこちらの記事にまとめています。
ImageJなどの画像解析ソフトを使って、コンピュータに数えてもらうのが一般的になってきています。
しかし...コンピューターは「人の目」ほど直感的に対象物を捉えることはできないのです。
コンピューターが対象物を捉えるためには、数値処理によって画像を分割して、(何かしらの)基準を設けて対象物と背景を判別させるという作業が必要になってきます。
この「基準を設けて対象物と背景を判別させる」という処理のことを「分節化(sgmentation)」と言います。
画像解析で一般的に使われている分節化は「輝度閾値(intensity threshold)」。
特定の輝度基準を設けて、それ以上の輝度を持つものを対象物であるとコンピューターに判断させる方法です。
今回は、「輝度閾値」を設定して画像中の対象物「核」を抽出してみます。
画像データは数値データって知っていましたか?下の記事に詳しくまとめています。

輝度閾値による分節化で核を抽出する
こちらの記事で使ったDAPI染色画像を使って、核の部分を抽出してみます。
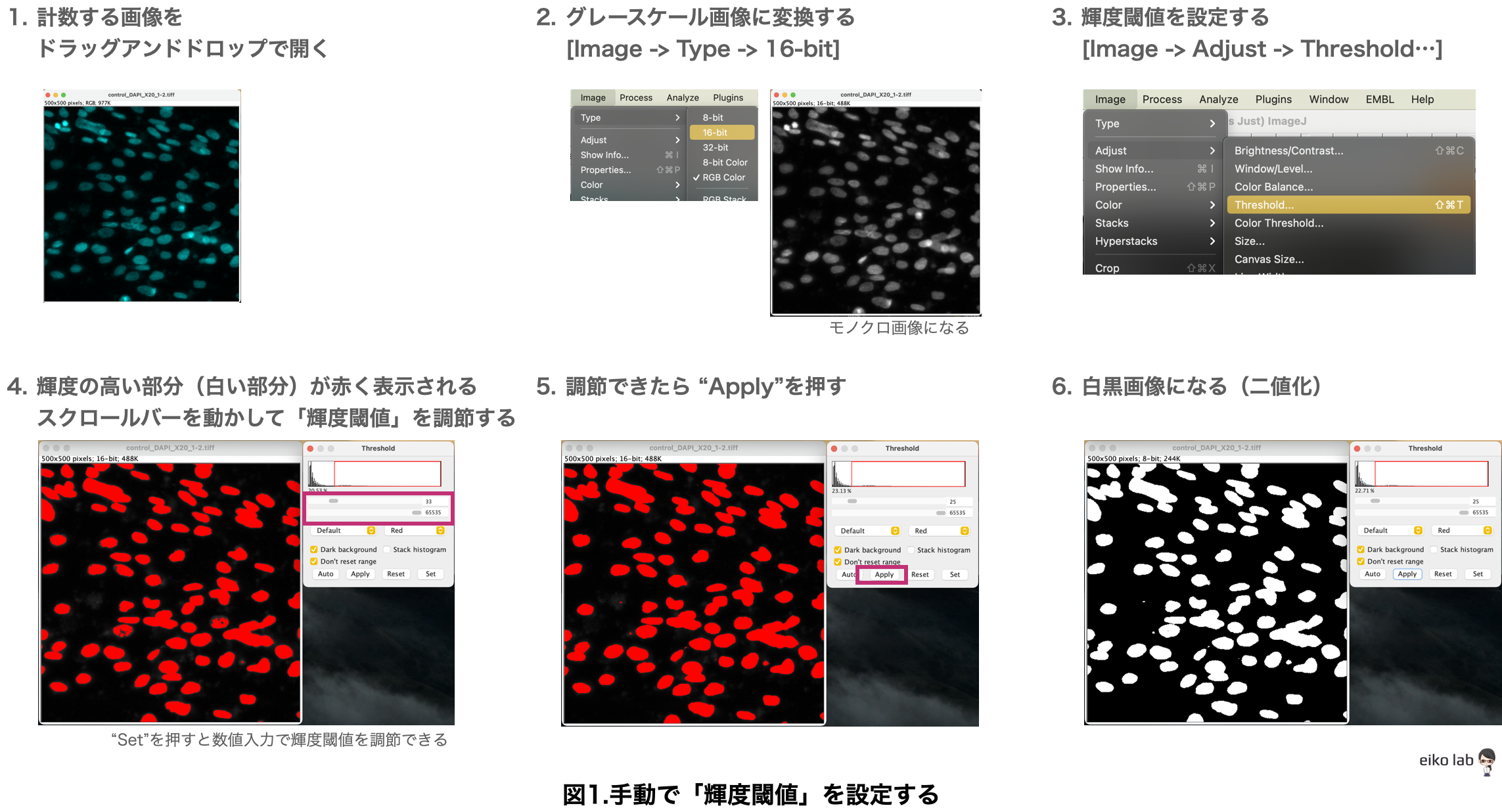
ImageJで「輝度閾値」を設定する時には"Threshold"を使います。
手でスクロールバーを調節して、目で見ながら「輝度閾値」を決めて、核の領域を判別させることができるようになるのです。
最適な輝度閾値を探す
手動で調節するの、めんどくさいなぁって思いませんでしたか?
毎回画像を見ながら、手動で輝度閾値を調整するのは、人の目で一個一個数えるのと同じくらい時間がかかります。
さらに人の目で毎回調整するのは、解析に一貫性がなく、客観性に欠けるというデメリットもあるのであまりおすすめできる方法ではありません。
実は、ImageJには「輝度閾値」を自動的に設定するアルゴリズムがいくつか搭載されているのです。
解析に一貫性を持たせるためにも、自動的に輝度閾値を設定してくれるアルゴリズムを導入するのがおすすめ。
このアルゴリズムを導入しておけば、マクロを組んで自動で解析してもらうという応用が効くので解析する画像が増えても怖いものなしです!
ImageJに組み込まれている輝度閾値設定用のアルゴリズムを一覧で見る方法を下の図にまとめました。

サンプルの画像では、「Li」が最適という結果でした。
このままImageJを使って白い領域の数を数えることはできるのですが、核がいくつか集まった部分は大きな一つの領域になっていたり、核ではない小さな領域が残ってしまっていたりしています。
計数の方法もImageJで設定していきましょう。
大きな領域を分割する、小さな領域を計数しない
核の部分は「輝度閾値」を設定することで、白い領域として抽出できました。
画像をよく見てみると、核がいくつかくっついていたり、小さな領域が残っていますよね。
これは「輝度閾値」ではどうしようもできないので、解析方法を色々設定することで解決していきます。
大きな領域を分割する
核がいくつかくっついている大きな領域については、ImageJの"Watershed(分水嶺)"アルゴリズムを使って分けていきます。
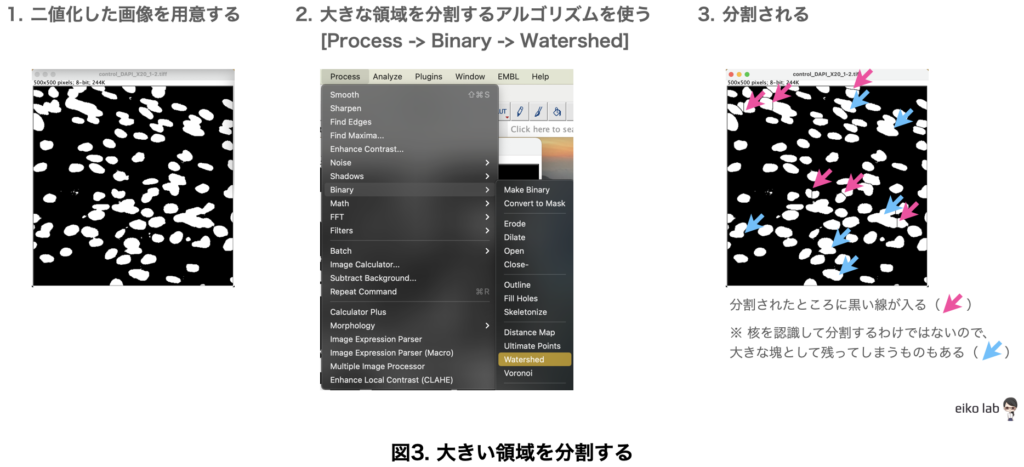
図3に示したように、うまく分割できるところとそうではないところが出てきてしまいます。
(Watershedのレベルを調節できれば良いのですが、今のところその方法は勉強中です。"Adjustable Watershed"というものがあるので、もしかしたらうまく分割できるようになるかもしれません。)
ImageJ "Watershed"を調節して核を数えてみる

小さな領域を計数しない
小さな領域を計数しないようにするために、下の図のような設定をして計数します。

小さい領域を数えないで、計数することができました。
【ImageJ】細胞の核の数を数える#2 まとめ
この記事の内容をまとめていきます。
- コンピューターは「ある数値基準」に従って分節化することで対象物をできるようになる
- ImageJの"Threshold ..."を使えば「輝度閾値」を設定することができる
- ImageJに組み込まれているアルゴリズムを使えば解析の客観性が保たれる
- 大きな領域を分割したり、小さい領域を取り除いたりして計数する
次の記事では、「マクロを組んで細胞の計数を自動化」していきます。
この記事に記載したImageJの使い方は下記の書籍を参考にしました。


















