
『【ImageJ】細胞を数えるシリーズ』では、ImageJで細胞を計数マクロを組んでいくことを目標としてやってきました。
第4弾で計数するマクロが完成し、第5弾でImageJ Fiji上で使えるようにしました。
第6弾の今回は、手動で計数した結果とマクロの計数結果を比較していきます。
せっかくマクロを組んで自動化しても、手動で計数したものと全然違うのであれば、マクロでやる意味はなくなってしまうので...
マクロを使って解析したデータをまとめる
作成したマクロを使って、出されたデータを見てみます。(私のパソコンにはExcelが入っていないので、「Numbers」を使ってcsvファイルを開いています)

写真ごとのcsvファイルを開いて、データを表に打ち込んでいきます。(地味に時間がかかります...)
組んだマクロのコードはこちら↓
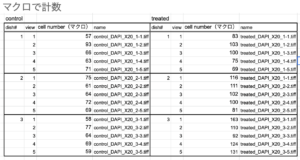
マクロで計数している様子はこちら↓
ちなみに、手動で計数したデータは...
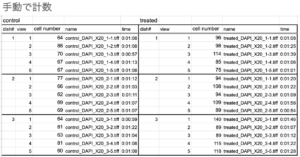
手動での計数の仕方はこちら↓
数値の羅列だけでは違いがあるのかどうかはよくわからないですよね。
手動のデータとマクロのデータを比較する
手動で解析したデータとマクロのデータを比較するために、Macでデフォルトで入っている表計算ソフト「Numbers」を使って平均値の棒グラフを簡単に書いていきます。
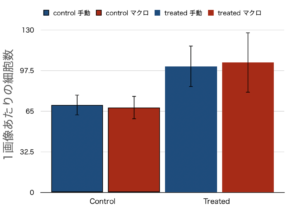
青いバーが手動、赤いバーがマクロで計数した結果。左側の二つがコントロール群、右側二つが薬剤添加群です。
エラーバーは標準偏差(SD)を示しています。
今回は解析結果のばらつき度合いを示したかったのでSDを使いました。
標準偏差と標準誤差の違いはこちらの記事にまとめています。
パッとみた感じは大きな違いはなさそうですね。
平均値のグラフだと、それぞれのデータの対応やデータのばらつき度合いが全くわかりません。
そこで生命科学の分野では、最近は箱ひげグラフやデータ一つ一つをプロットすることが推奨されています。
有意な差があるのかどうかを調べるために、「Numbers」でもできるt検定(両側検定、対応のある2群の検定)をして見ると、controlの場合はp値が0.117、treatedの場合はp値が0.360でどちらも帰無仮説を棄却できず、有意な差はないという結果を得られました。
t検定についてはこちらの記事にもまとめています。
今回はデータ量がそこまで多く無かったので手で入力して簡単にデータの結果を見ることができましたが、データ量が多くなってくるといちいちファイルを開いてデータを確認するのはめんどくさいですよね。
次回からは、Rを使ってデータを読み込んでグラフを作って検定するを自動化していく「Rを使う」シリーズを始めていこうと思っています。



















