
DNA(核酸)サンプルを定量する時、あなたはどうしますか?
生命科学系の研究室では、DNA(核酸)のサンプルを扱うのは日常茶飯事。
何も考えずにナノドロップに向う人も多いのではないでしょうか?
では、エタノール沈殿ではなくPCRで増幅させたDNAサンプルはどうやって濃度を測定していますか?
何も考えずにナノドロップ行きでしょうか?
この記事では、DNA(核酸)の定量法の原理について、PCR産物はナノドロップで測定できない理由などをまとめていきます。
※DNA(核酸)と表記しているのは、測定の対象としてはDNAが多く用いられますが核酸の中でもRNAも測定可能であることを示しています。RNAもDNAと同様にナノドロップで定量が可能です。
DNA(核酸)を定量する方法
DNA(核酸)の濃度を定量方法は大きく、「蛍光分析法」と「吸光度法」に分けられます。
「蛍光分析法」では、DNAの二本鎖の中に入り込んで蛍光を発する試薬を使用して蛍光の強さからDNAを定量する方法です。
エチジウムブロマイドやSYBR系の試薬を使ってアガロースゲル中のDNAを光らせるのも「蛍光分析法」の一種です。
アガロースゲル電気泳動についてはこちらの記事をご覧ください。

また、定量PCR(quantitative PCR)やDNAマイクロアレイなども「蛍光分析法」が使われています。
定量PCRについてはこちらの記事をご覧ください。

もう一つの「吸光度法」は、ナノドロップなど研究室で日常的に使われているDNA定量法です。
「吸光度法」の原理
「吸光度法」とは?
「吸光度法」とは、特定の波長を物質に当てた時に吸収された光の割合を測定する方法です。
ある物質に光を当てて(入射光)を当てて、物質を通過して出てきた光(透過光)がどれくらい弱まったか?(吸光度)を評価します。
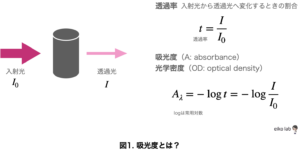
「吸光度法」で物質の濃度を測定する
この「吸光度」は、光が通過した物質の長さ(光路長)と物質の濃度に比例します(ランベルト・ベールの法則)。
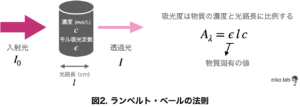
光路長を一定にして、吸光係数がわかっている物質であれば、吸光度から濃度の測定が可能になります。
また、吸光係数がわからない場合は濃度がわかっているもの(標準液)を使って検量線を引いて、濃度未知の試料の測定を行います。
吸光度はなぜ”-log10”で表すのか?
吸光度はなぜ、「入射光ー透過光」の値そのものではなく"-log10"を付けるのでしょう?
"-log10"の使用を導出する方法があるのですが、ちょっと難しい。
ここでは割愛して、"-log10"を使うとどう言うことが起こるのか簡単にまとめておきます。
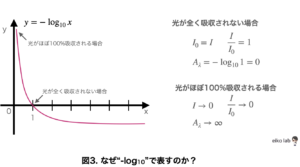
ランベルト・ベールの法則から吸光度を算出する詳しい計算過程はこちらにまとめています。

吸光度法を使ってDNA(核酸)を定量する
「吸光度法」を使って核酸を定量する方法はいつ頃から使われ始めたのでしょうか?
最初は「DNAを定量する」という使われ方ではなく、DNA(核酸)のコンタミの程度を調べる方法として用いられていたようです。
1940年代はじめ、カイザー・ヴィルヘルム研究所のオットー・ワールブルクは解糖系の酵素の一つであるエノラーゼ(ホスホピルビン酸ヒドラターゼ)の単離を試みていました。
その当時、タンパク質を単離する時にはどうしても核酸がコンタミ(混入)してしまうことが頻繁に起こりました。
タンパク質の吸光のピークは280 nmですが、核酸も280 nmの光を吸収してしまいます。
そこで核酸の吸光のピークである260 nmでの吸光度との比を取ることで、単離したタンパク質にどれくらい核酸がコンタミしているかの指標とすることにしました。
1942年のエノラーゼの単離に関する論文に、どれくらい核酸がコンタミしていると260 nmと280 nmの吸光度の比がどうなるか記載されています。
「タンパク質の核酸のコンタミの指標」から「核酸の濃度・純度の指標」と言う逆の使われ方をし始めたのは、それから10年以上経った1950年代後半からとされています。
吸光度法を使ったDNA(核酸)の定量
核酸は260 nm付近の波長で吸光のピークを持ちます。
核酸の構造の中でも「塩基」の部分が260 nm付近に吸光のピークを持つのですが、「塩基」は、アデニン・シトシン・グアニン・チミン(・ウラシル)によって構造が微妙に異なります。
それぞれの塩基の吸光ピークは次の表の通りです。

ばらつきはありますが、一般的に260 nmが使われます。
核酸の濃度を調べるためには、核酸の吸光定数aを知る必要があるのですが塩基の種類・核酸の長さ・配列によって吸光定数が変化してしまうため厳密には決められません。
そこで一般的な吸光度計(ナノドロップ)では吸光定数の代わりに、260 nmの吸光度(A260)が1となる核酸濃度を使って、測定値を濃度に変換しています。

DNA(核酸)の純度について
純度の高い核酸の吸光度を、横軸に波長をとってプロットすると下記の図のようになります。
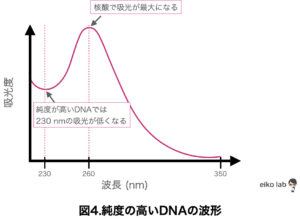
核酸の純度の指標となるのがA260/A280の比。
260 nmは核酸、280 nmはタンパク質の吸光が最大となる波長です。
この比を取ることによって、核酸中のタンパク質の混入度合いを評価することができます。
一般的に、DNAは1.8程度、RNAは2.0程度で核酸の純度が高いとされます。
A260/A280の値が小さい場合、タンパク質やフェノールの混入率が高いと判断されるので精製をやり直したほうが良い場合が多いです。
DNAサンプルのA260/A280の値が1.8よりも高い場合は、RNAのコンタミが疑われます。
ちなみに、最近はA260/A230の値も純度の判定に用いられるようになっています。
230 nmの波長は主にフェノール、エタノール、グアニジンチオシアン酸などが吸光します。
A260/A230の値が1.8よりも低い場合は、核酸抽出過程でのバッファーの洗いが足りない可能性が高いです。
PCR産物がナノドロップで測定できない理由
最後に、PCR産物が吸光度法(ナノドロップ)で測定できない理由をまとめましょう。
PCR産物にはどんなものが含まれているのか、整理してみます。
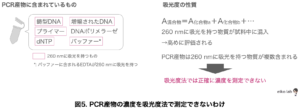
吸光度の特徴から、混合物の吸光度はそれに含まれる化合物の吸光度の総和になります。
PCR産物には260 nmに吸光を持つ化合物が複数混ざり合っているため、測定したいPCR産物の実際の濃度よりも高めに評価されてしまい、正確性を失ってしまいます。
PCR産物を正確に測定したい場合は、一度アガロースゲル電気泳動などで混合物(プライマーやdNTPなど)を分離して、バンドを切り出して目的物だけにして測定するのが一般的です。
この過程を切り出し回収と呼びます。
PCRの基本的な原理についてはこちらの記事にまとめています。

アガロースゲル電気泳動に関してはこちらの記事

アガロースゲルからの切り出し回収に関してはこちらの記事をご覧ください。

DNA(核酸)を定量する まとめ
DNA(核酸)の定量についてまとめます。
- 核酸を定量する方法は「蛍光分析法」と「吸光度法」がある
- 「吸光度法」にはナノドロップが一般的に用いられる
- 核酸の中で塩基の部分が260 nmの波長の吸光を持つ
- 核酸の純度の指標としてA260/A280の値がよく用いられる
- PCR産物の濃度はナノドロップで測定できない
参考文献














